ChIC/CUT&RUN Assay Kit概览
Related Products:
- CUT&RUN-validated antibody: For best results with your samples
- CUT&RUN Spike-In Control: Evaluate CUT&RUN assay datasets confidently for comparison
CUT&RUN(Cleavage Under Targets & Release Using Nuclease)是一种研究各种染色质相关蛋白及其修饰的全基因组分布的表观遗传学方法,是ChIC(chromatin immunocleavage)的衍生方法1-3。 类似于ChIP(chromatin immunoprecipitation),CUT&RUN也是利用抗体靶向染色质相关修饰和蛋白,但其所需细胞量和测序深度比传统ChIP更低。
在CUT&RUN中,抗体靶向目标蛋白从而结合在完整细胞中的染色质上,然后微球菌核酸酶(MNase)在目标蛋白结合位点特异性地剪切DNA,所释放的DNA片段被纯化、测序并定位到参考基因组序列上,就可推断出蛋白在DNA上的结合位点。ChIP需要将蛋白与DNA交联,这一步可能导致假阳性,而CUT&RUN不需要交联反应。
作为研究染色质相关蛋白的极佳工具,CUT&RUN灵敏、特异性高且所需细胞量比ChIP更少,是研究转录因子等染色质相关蛋白的结合方式和全基因组的组蛋白修饰的理想方法。染色质相关蛋白在调控诸如基因表达、DNA复制、DNA修复和细胞分化等过程中发挥很重要的作用,研究这些蛋白的结合方式可以为这些细胞活动的调控提供潜在思路。
参考文献
1. Schmid, M. et al. Mol Cell, 16(1): 147-157 (2004)
2. Skene, P.J. et al. (2017) Elife 6, e21856
3. Skene, P.J. et al. (2018) Nat. Protoc., 13, 1006-1019
ChIC/CUT-RUN Assay Kit的优势:
- 适用于5,000-500,000个细胞的样品
- 提供完整的试剂和优化方案
- 适用于全基因组染色质相关蛋白分析
ChIC/CUT-RUN实验流程

CUT&RUN, CUT&Tag与ChIP-Seq的对比
| CUT&RUN | CUT&Tag | ChIP-Seq | |
|---|---|---|---|
| 是否在非变性条件下进行 | 是 | 是 | 否 |
| 染色质片段化方法 | MNase酶切 | Tn5的转座反应 | 超声 |
| 所需细胞数量 | 50万 | 0.5-50万 | 100-1000万 |
| 所需测序深度 * | 8M † | 2M ** | 20-50M |
| 是否整合建库过程 | 否,需要另外的建库步骤 | 是,通过片段化加adaptor | 否,需要另外的建库步骤 |
| 适用的靶点 | 广泛适用于组蛋白修饰、转录因子与辅因子 | 主要适用于组蛋白修饰、少许转录因子与辅因子 | 广泛适用于组蛋白修饰、转录因子与辅因子 |
| 所需时长 | 1-2天 | 1-2天 | 2-3天 |
* Kaya-Okur et al. Nature Communications (2019) 10:1930
** 对于丰度较低的靶点,建议测序深度8-10M reads
† 对于转录因子,建议测序深度25M reads
ChIC/CUT-RUN Assay Kit组分:
ChIC/CUT-RUN试剂盒各组分在两种温度下运输:一盒为需-20℃储存的试剂,通过干冰装运;另一盒为不需冻存的试剂,需于4℃储存,且在室温下运输。请依据试剂盒说明书对试剂盒中各组分进行合理保存,所有试剂在正确温度下储存可保证6个月内稳定。
- Protease Inhibitor Cocktail, 500 µl, -20°C储存
- ChIC/CUT&RUN pAG-MNase, -20°C储存
- Glycogen 20 mg/ml, -20°C储存
- Histone H3K4me3 pAb, 10 µg, 1 mg/mL, -20°C储存
- Negative Control IgG, Rabbit, -20°C储存
- 1 M Spermidine, -20°C储存
- Nuclei Isolation Buffer, 4°C储存
- Concanavalin A Beads, 4°C储存
- 0.1 M CaCl2, 4°C储存
- Stop Solution, 4°C储存
- 1X Binding Buffer, 4°C储存
- Dig-Wash Buffer, 4°C储存
- 0.5 M EDTA, 室温储存
- DNA Purification Wash Buffer, 室温储存
- DNA Purification Elution Buffer, 室温储存
- DNA Purification Binding Buffer, 室温储存
- 3M Sodium Acetate, 室温储存
- DNA Purification Columns SF, 室温储存
- 0.2 mL Strip Tubes, Attached Caps, 室温储存
- RNase A, -20°C储存
- 5% Digitonin, -20°C储存
ChIC/CUT&RUN数据展示
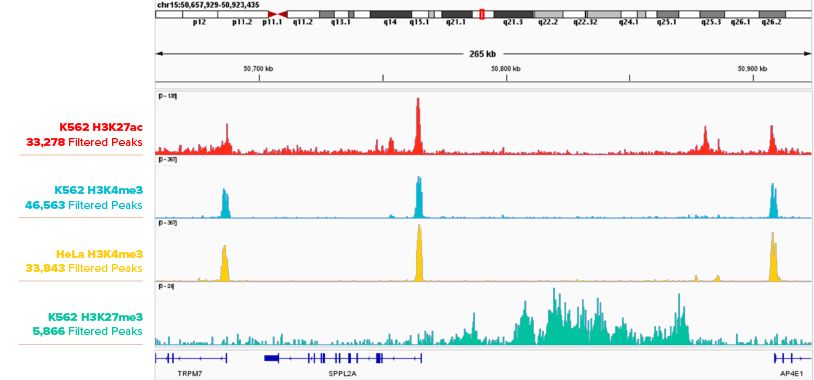
图1: CUT&RUN显示K562和HeLa细胞中的组蛋白修饰
使用CUT&RUN对500,000个K562细胞和500,000个HeLa细胞中的组蛋白修饰进行检测,数据可视化在基因组浏览器IGB中。所用到的抗体:H3K27ac: Cat. No. 91193, H3K4me3: Cat. No. 91263, H3K27me3: Cat. No. 39155。
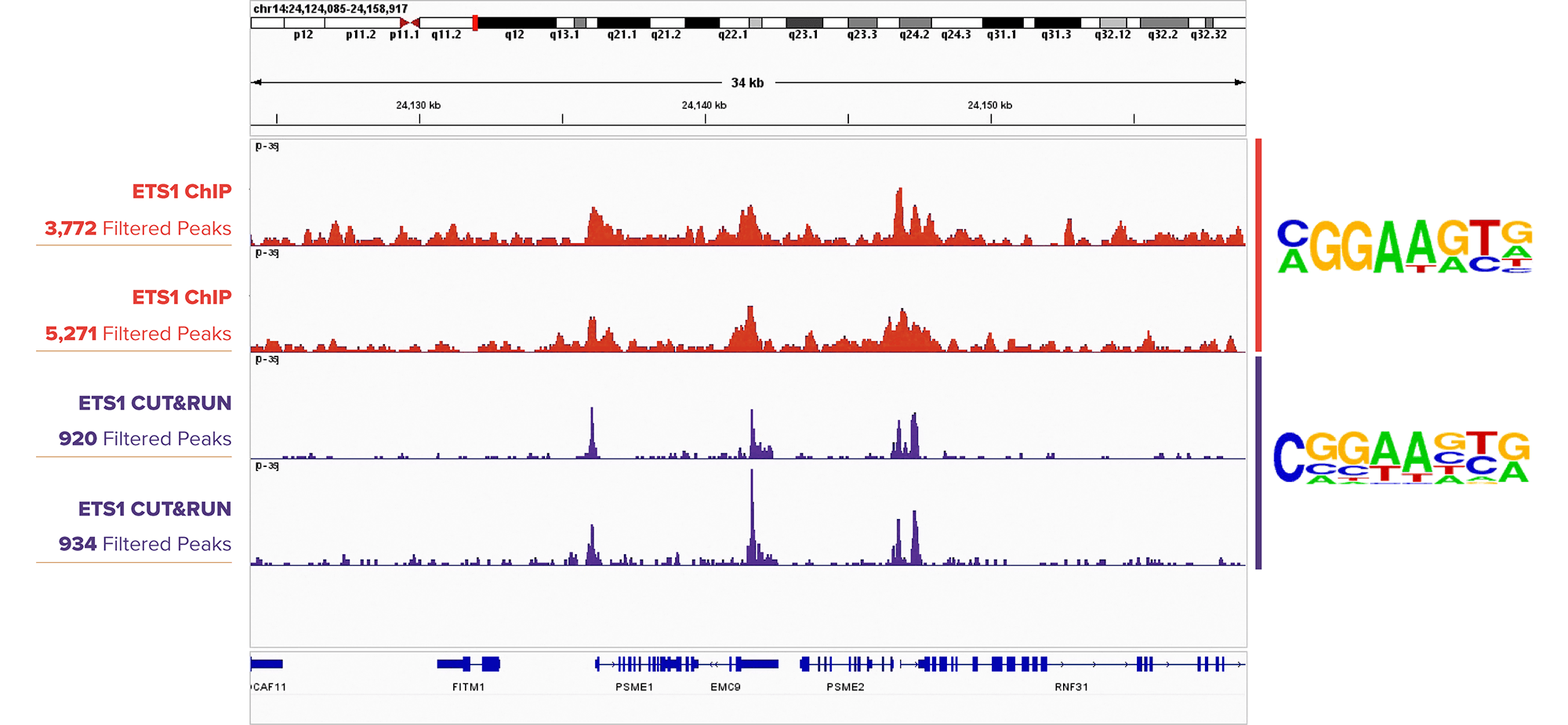
图2: 使用ETS1抗体进行CUT&RUN和ChIP-Seq实验得到相似的结果。
使用ETS1抗体在DIPTG细胞中进行ChIP-Seq实验(上图左第1,第2栏)与在K562细胞中进行CUT&RUN实验(上图左第3,第4栏),数据可视化在基因组浏览器IGBV中。来自 Homer 分析的序列分析(上图右)显示,在两组样本中,ETS1 motif均富集。所用到的抗体:ETS1: Cat. No. 39580。
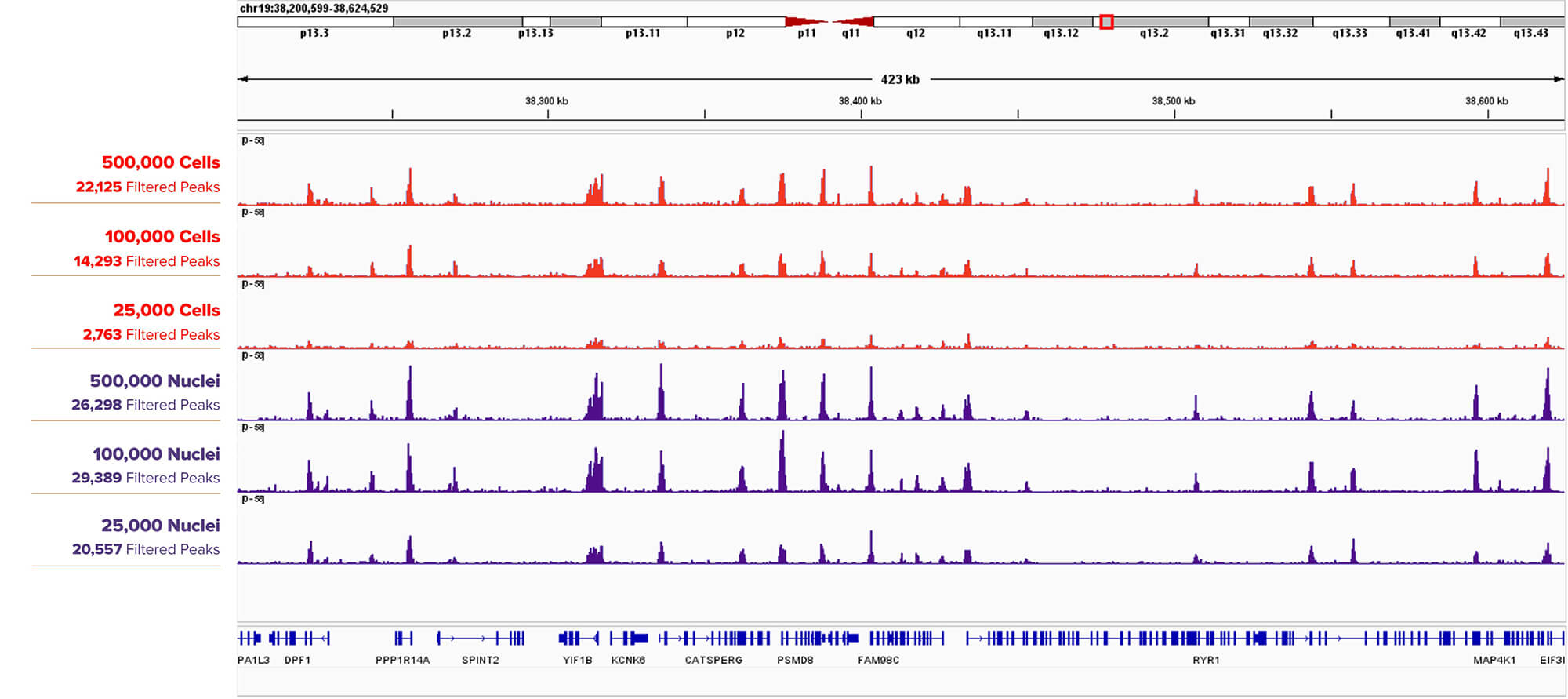
图3: CUT&RUN 检测比较等量K562 细胞与细胞核中H3K4me3 修饰结果。
CUT&RUN检测500,000, 100,000, 25,000个K562细胞(第1,第2,第3栏)或细胞核(第4,第5,第6栏)中H3K4me3 修饰,在更低的input下,细胞核样品仍呈现比细胞样品更高的峰值。所用到的抗体:H3K4me3: Cat. No. 39060。
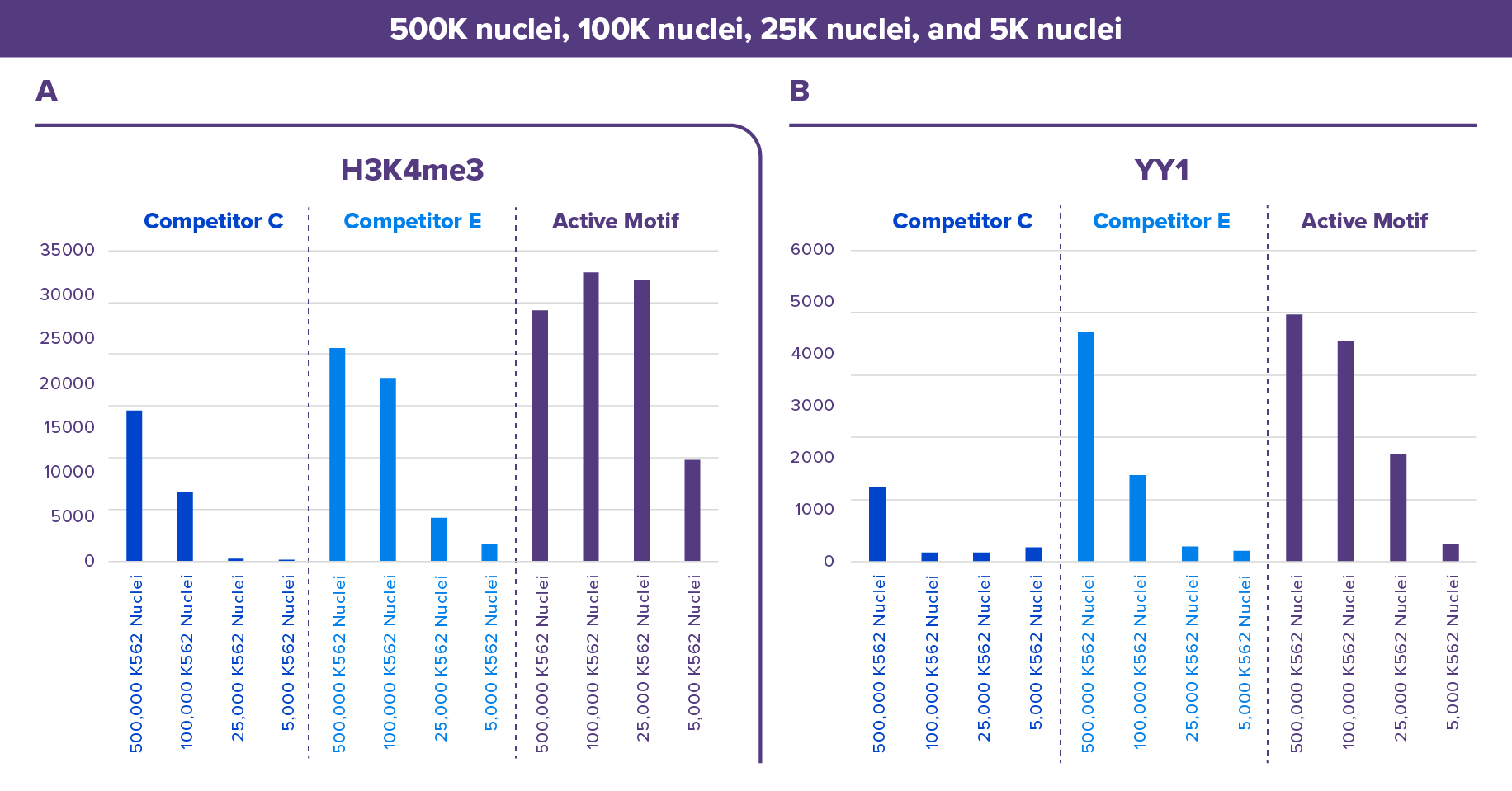
图4: Active Motif的CUT&RUN试剂盒显示出比竞争对手试剂盒更好的结果。
分别使用Active Motif,竞争对手C或竞争对手E的CUT&RUN试剂盒检测500,000, 100,000, 25,000个K562细胞核的YY1和H3K4me3信号。所用到的抗体:YY1:Cat. No. 61980,H3K4me3: Cat. No. 39916。
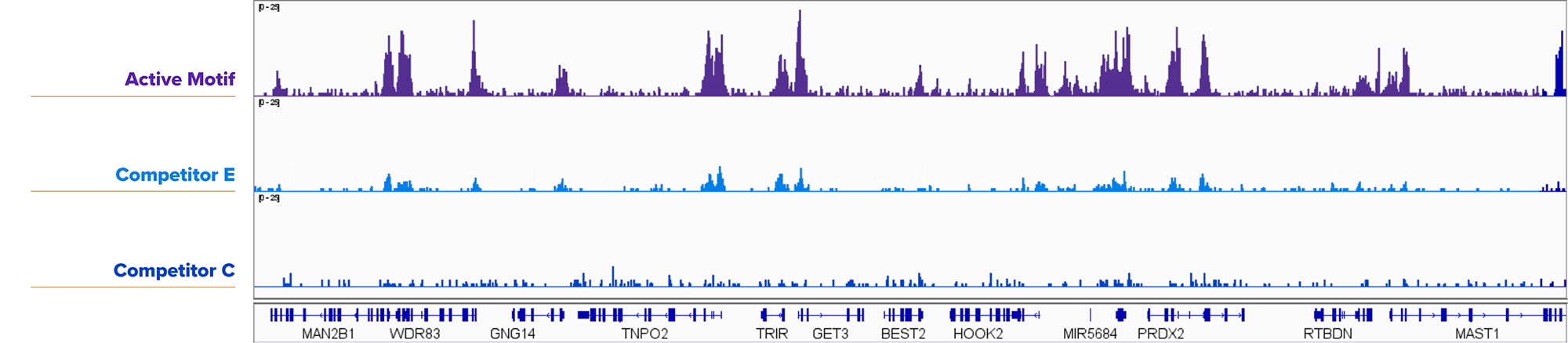
图5: Active Motif的CUT&RUN试剂盒检测5,000个细胞核的组蛋白修饰结果优于竞争对手试剂盒。
分别使用Active Motif,竞争对手E或竞争对手C的CUT&RUN试剂盒检测5,000个新鲜的K562细胞核的H3K4me3信号,Active Motif的CUT&RUN试剂盒得出的结果比竞争对手更可信。所用到的抗体:H3K4me3: Cat. No. 91263。
图6: Active Motif的CUT&RUN试剂盒检测25,000个细胞核的转录因子结果优于竞争对手试剂盒。
分别使用Active Motif,竞争对手E或竞争对手C的CUT&RUN试剂盒检测25,000个新鲜的K562细胞核的YY1信号,Active Motif的CUT&RUN试剂盒得出的结果比竞争对手更可信。所用到的抗体:YY1: Cat. No. 61980。
ChIC/CUT&RUN试剂盒常见问题
看不见细胞核沉淀怎么办?
沉淀可能是看不见的,确保带盖子的离心管在离心机中朝向是确定的,这样才能知道沉淀应该处于离心管的什么位置,并且在之后的操作中也需要小心。
推荐的测序深度是多少?
对于转录因子,我们推荐测序深度为25M reads;对于一些组蛋白修饰,测序深度达2M reads即可。
What is the recommended read length for CUT&RUN?
The recommended read length for CUT&RUN is PE38.
检测转录因子需要的细胞量是多少?
我们推荐500,000个细胞用于CUT&RUN实验,且需使用已被验证可用于CUT&RUN的抗体。
为什么数据的背景太高?
确保一开始使用的是活细胞,可以使用以下方法检查细胞是否受损:Coutess II等自动细胞计数方法,或台盼蓝染色后用血细胞计数器计数。
阴性对照可以产生文库吗?
阴性对照IgG可以产生文库。
SPRI beads没有办法沉淀或者沉淀所需时间太长,怎么办呢?
首先用磁条将SPRI beads聚集在离心管的顶部,然后逐渐向离心管的底部移动,最后把管子放在磁力架上。
Active Motif的哪些抗体已被发表用于CUT&RUN?
以下列出的抗体已被发表用于CUT&RUN,但这些抗体是否可用于CUT&RUN还没有被Active Motif验证。
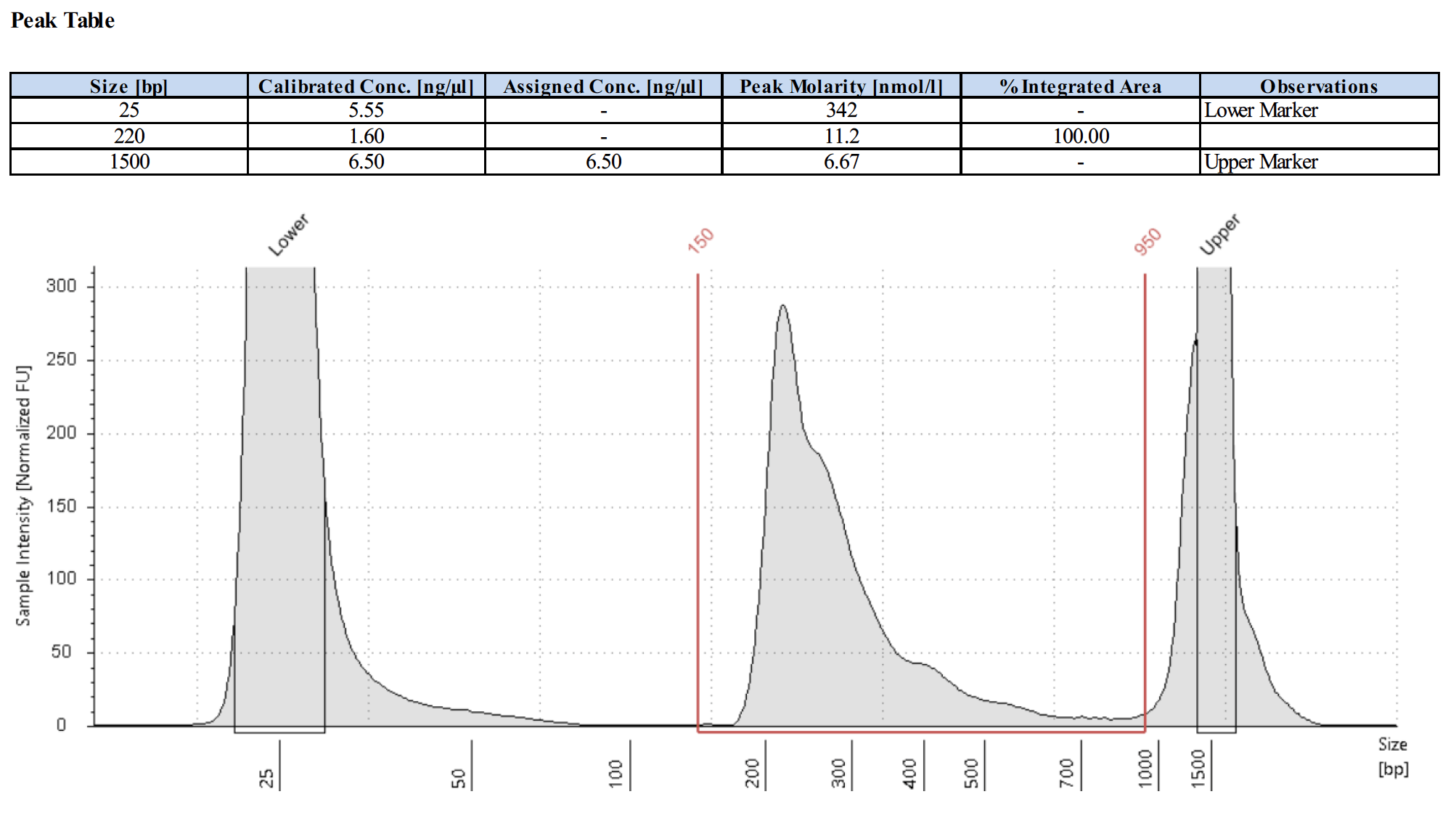
阳性对照抗体的测序峰图应该是怎样的?
下面为4个阳性对照的测序峰图。




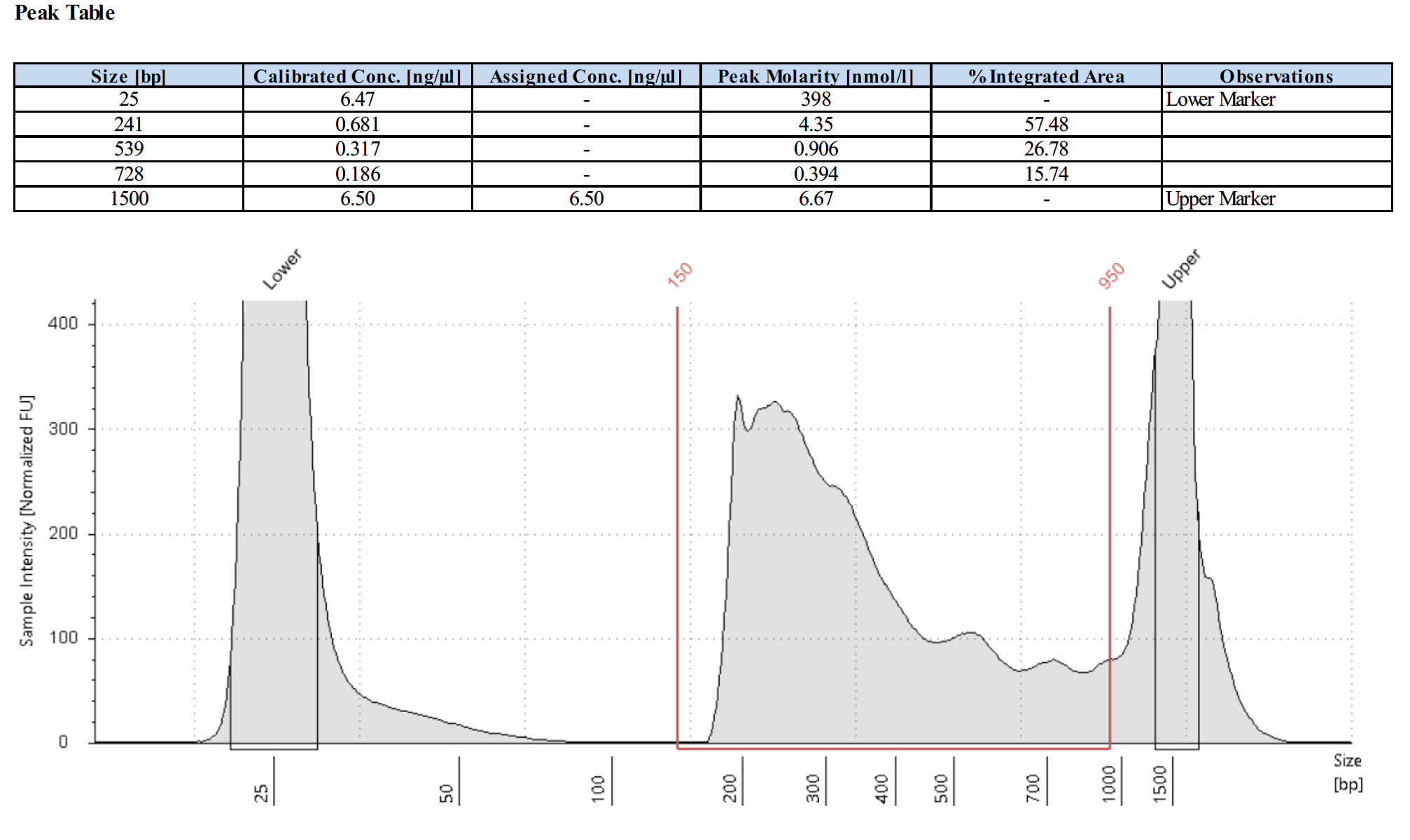
阴性对照IgG的测序峰图应该是怎样的?
下面为2个阴性对照的测序峰图。


推荐使用细胞样品还是细胞核样品进行CUT&RUN实验?
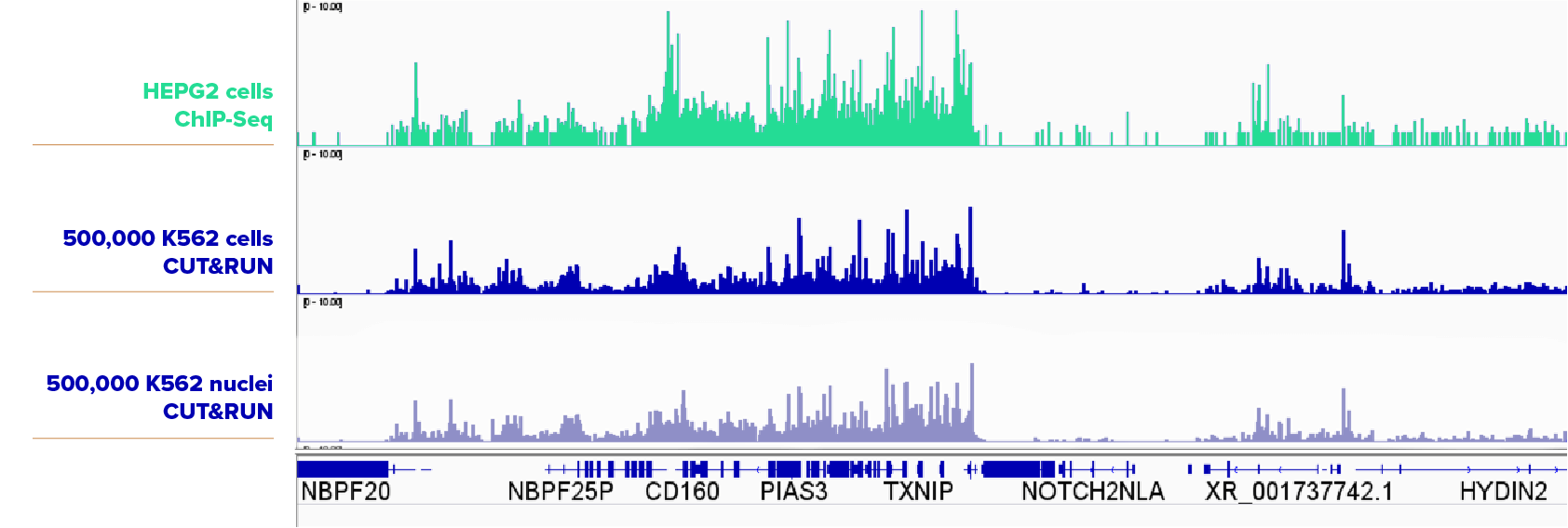
我们推荐用细胞核进行实验,但是在有些情况下,靶向转录因子的细胞样品可得出更高的峰,详情见下图。
CUT&RUN检测500,000个K562细胞和500,000个K562细胞核中的SUZ12,细胞样品和细胞核样品得出的结果相似,第一栏是HEPG2细胞的ChIP-seq结果作为对照。所用到的抗体:SUZ12: Cat No. 39357。
CUT&RUN检测500,000个K562细胞和500,000个K562细胞核中的BRD2,BRD3或BRD4。对于BRD2和BRD3,细胞样品的峰稍高于细胞核样品;对于BRD4,细胞核样品的峰高于细胞样品。所用到的抗体:BRD2: Cat No. 61797,BRD3: Cat No. 61489,BRD4: Cat No. 91301。
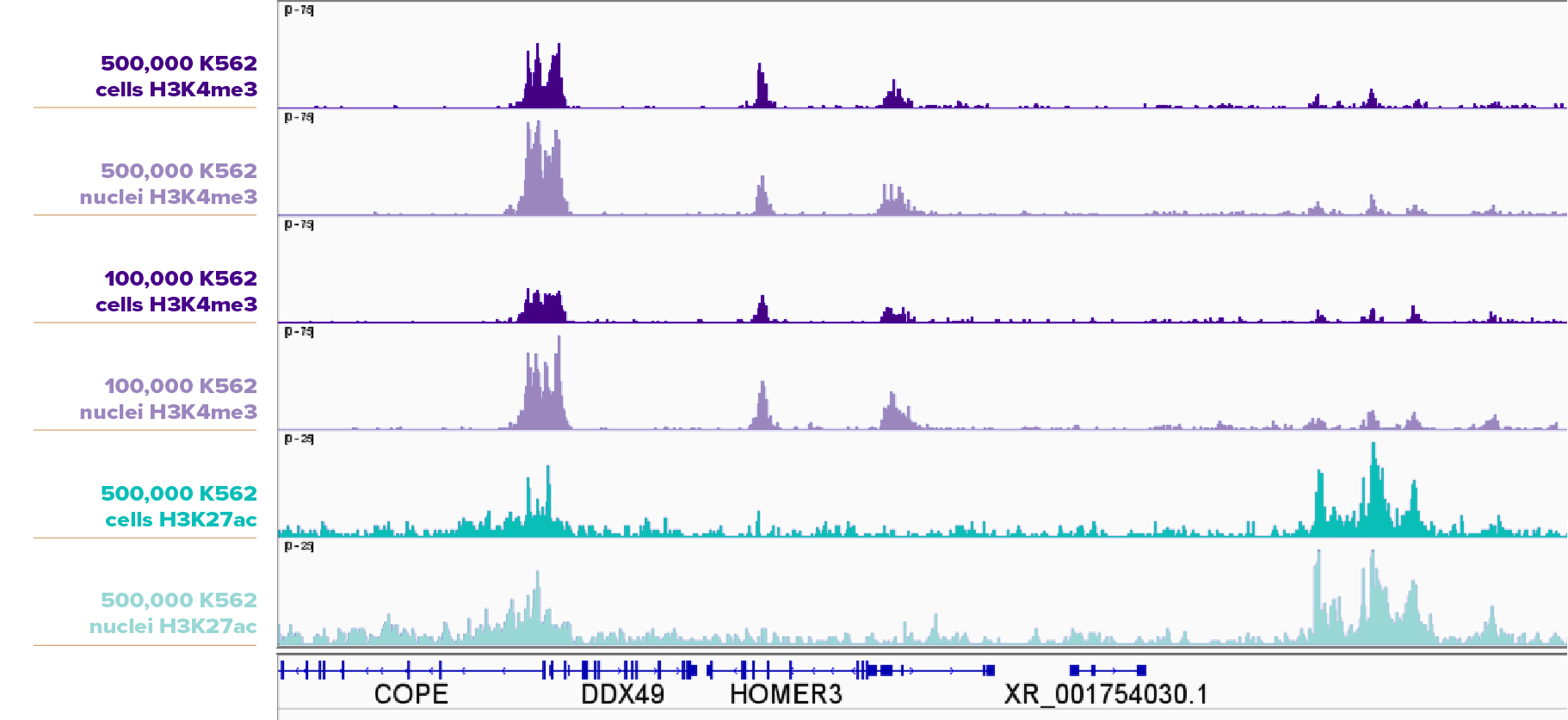
CUT&RUN检测500,000个和100,000个K562细胞或细胞核中的H3K4me3,以及CUT&RUN检测500,000个K562细胞或细胞核中的H3K27ac,细胞核样品的峰高于细胞样品。所用到的抗体:H3K4me3: Cat No. 91263,H3K27ac: Cat No. 91193。
ChIP-Seq或者CUT&Tag验证的抗体可以用于CUT&RUN Assay Kit吗?
CUT&RUN,CUT&Tag和ChIP-Seq实验流程不一样,如果某种抗体适用于CUT&Tag或者ChIP-Seq,不代表这款抗体也适用于CUT&RUN,我们建议使用我们CUT&RUN验证抗体列出的抗体进行CUT&RUN。
What does the library peak profile look like for YY1?
What does the library peak profile look like for JunD?
ChIC/CUT&RUN Assay Kit文件